マルチオミクス解析の発展その2 ...-ICT技術調査 デジタルヘルス情報提供-株式会社テックナレッジハウス
 BLOG
/ デジタルヘルスの未来
BLOG
/ デジタルヘルスの未来
マルチオミクス解析の発展その2 – DNAのLLM Evo2の登場 –
ヘルスケア分野へのAIの活用は、オミクス解析がマルチオミクス解析へ進むと一段と進む、ということで、前回から具体的な研究やAIモデル開発の紹介をしている。
前回は、山梨大学をコホート研究でDNAのみでなくプロテオミクスも包含した研究を進めていると紹介をした。今回は、今年の2月に発表された世界最大規模のゲノム解析用の大規模言語モデル(LLM)のEvo2を紹介したい。米国のArc Instituteとスタンフォード大学が中心になって開発した。DNAの解析モデルだが、RNAやタンパク質の洞察まで行い、対象も人間は勿論真核生物全般、細菌、アーキアまでと範囲が広い。
IT家の私は、2025年3月にアメリカの西海岸サンノゼで開催されたNvidiaの年次技術コンフェレンスであるGTC 2025のヘルスケアのセッションをアーカイブのオンデマンドでみていたら、最先端のゲノム解析のための大規模言語モデル(LLM)のEvo2がNvidiaのバイオインフォマティックス用フレームワークのBioNeMo上で動作するようになり、オープンソースとして提供されるという説明があった。これは実際には2025年の2月19日に最初に発表されている。
バイオインフォマティックスやその周辺の勉強や研究をされている方々には説明の必要はないのだろうが、このEvo2というLLMとゲノム解析用LLMの現状につき簡単にここで触れたい。
(1)ゲノムに特化した大規模言語モデル(LLM)のEvo2
Evo2は、バイオインフォマティックス分野の大規模言語モデル(DNA配列を言語として扱う)として、HyenaDNA、Evoを経て、この分野では少なくとも発表時点で世界最大の次世代LLMである。Evoと比較して、大きくなったパラメータ数(7B⇒47B)やコンテキスト長(0.13M⇒1M)や充実した学習データ(15Kの有核生物のゲノムと113の原核生物のゲノムからの8.85兆のヌクレオチド)によりEvoでは対象が原核生物のみだったが真核生物に拡大された。
開発したのは、非営利のバイオインフォマティックス研究団体のArc Instituteとスタンフォード大学がリードするチームである。
Evo2を利用することで、図1の様に、DNA、RNA、タンパク質という3つの生体分子、および生命の3つすべての領域(真核生物、原核生物、古細菌(アーキア)*)にわたる諸洞察を得ることができる。
*: 生命の3領域である真核生物、原核生物、古細菌(アーキア)
この分類では、原核生物は細胞(バクテリア)とうことになる。原核細胞に古細胞を含めて真核生物と原核生物の2つに分類することもあるようである。
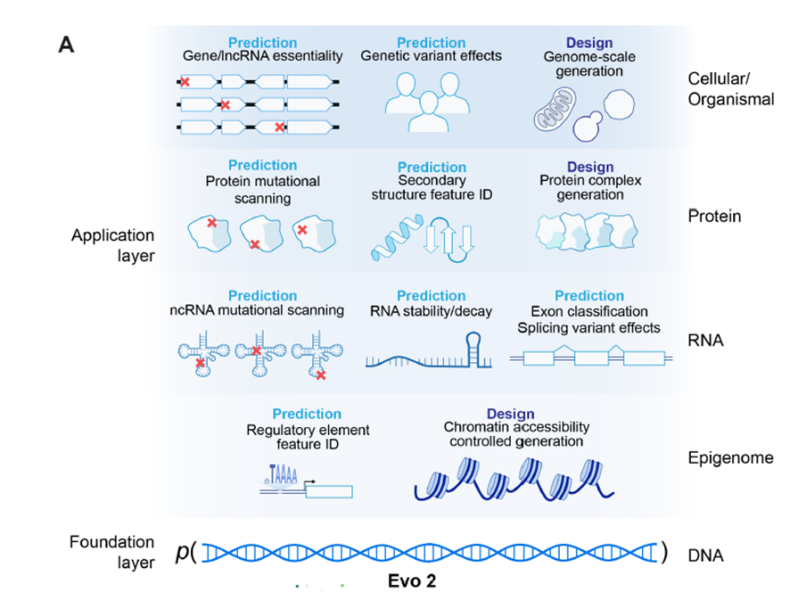
(図1)Evo2が適用できると思われるアプリケーションの分野
出展:”Genome modeling and design across all domains of life with Evo2”
このEvo2が出現したことにより、創薬や病気予測の更なる発展だけでなく、合成生物学など新しい分野の可能性を開く。
100万個の塩基レベルの超長文のコンテキストを扱えるので、従来のモデルでは見落とされてきた遠く離れたゲノム領域間の相互関係の把握も可能となった。AlphaFoldやESMFoldのように1つのタンパク質分子の立体構造や機能予測だけでなく、ゲノム全体をみて複数遺伝子や調節領域の相互作用までをモデルで扱える。
Evo2の応用分野としてはまず疾患バリアント診断への適用がある。
これまでも病気の予測はゲノム解析も使い行われてきたが、このEvo2では、タンパク質の機能や生物のフィットネスに影響を与える遺伝的変化を特定する汎用性を持っている。例えば、乳がん関連遺伝子BRCA1 の変異を用いたテストでは、Evo 2は、どの変異が良性か潜在的な病原性かを90%以上の精度で予測した。(詳細説明は文末の参考情報の3.のp.9にある)このような洞察は、ヒトの病気の遺伝的原因を発見し、新薬の開発を加速することによって、細胞実験や動物実験に必要な数え切れないほどの時間と研究費を節約することができる。
Evo2の2つ目の応用分野として、DNA配列そのものを生成があり、Evo2は非常に優れた生成能力を持っている。ミトコンドリア全体や細菌ゲノムレベルの長さでも、自然な構造を保ちつつ新規配列を自動で生み出せる。これは合成生物学の分野で特に注目されており、大規模な配列デザインやターゲットとする微生物の改変、新素材の開発などにつながると期待できる。
また、エピゲノム(遺伝子発現の制御機構)を考慮した配列生成も可能で、特定の領域を“開かせる”か“閉じる”かといった柔軟な設計要求に合わせて、何千塩基単位のDNAを自在に生成できる点も素晴らしい。
一方、巨大なパラメータをもつEvo 2の“ブラックボックス”を解き明かそうとする取り組みも進んでいる。専門領域の言葉を借りると、モデルの内部表現を細かく分析することで、タンパク質の二次構造やプロモーター、さらにはバクテリオファージ由来の可動要素などがどのように符号化されているのかが明らかにできつつある、ということになる。
こうした解析は、ゲノム理解の深化だけでなく、未知の遺伝子機能や生命現象の新たな発見につながる可能性がある。
潜在的な倫理的リスクと安全性を考慮して、Evo2は基本データセットからヒトや他の複雑な生物に感染する病原体を除外し、これらの病原体に関するクエリに対してモデルが回答を返さないようにしているという。
Evo2の詳細は、文末の参考情報の論文3.とこの論文の日本語でのポイント解説の4.を参考にされたい。
(2)生物情報学での大規模言語モデル(LLM)
バイオインフォマティックス分野でのLLMを整理した論文はいくつかある。最新だとたとえば、Large Language Models in Bioinformatics: A Survey (香港中文大学のZhenyu Wang他の著、2025/5、ACL2025で発表)。
バイオインフォマティックス分野のLLMは、大きく分類するとすると、DNA、RNA、タンパク質、シングルセルRNA(scRNA)の4つの分野になる。
下の図2は、この論文に掲載されている図で、バイオインフォマティックスの代表的なLLMを時系列に整理し、この4つにうちのどれに属するか、及び、LLMの構造の分類(エンコーダか、デコーダーか、エンコーダとデコーダを両方備えるか)を整理している。
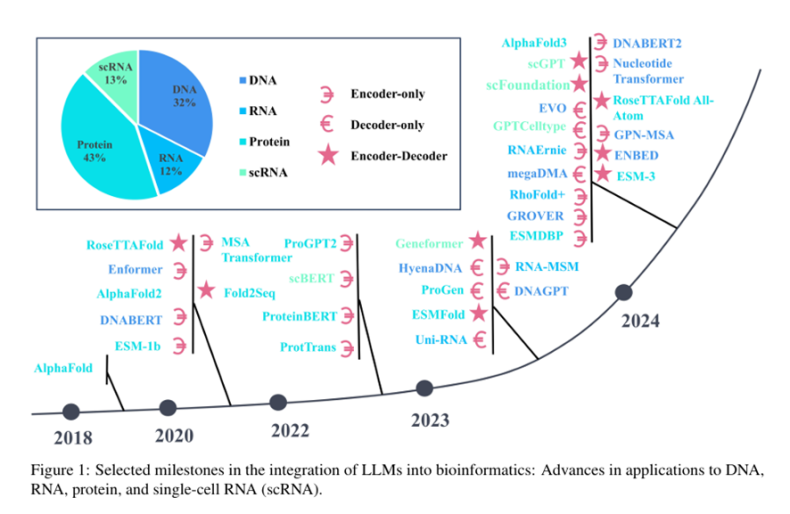
(図2)バイオインフォマティックスのLLMの歴史
出展:Large Language Models in Bioinformatics: A Survey
たとえば、既出で1つのタンパク質分子の立体構造や機能予測を行う、AlphaFoldは2018年、その後継はAlphaFold2が2020年、AlphaFold3は2024年に世に出、ESMFoldは2023年に出ている。
次に説明するEvo2の前身のHyenaDNAは2023年に、EVOは2024年に出ている。
(3)HyenaからEvo2への発展経緯
Evo2は、前述のように、2023年に発表されたDNA解析のLLMであるHyenaDNAをベースとして利用して、Evoを経て、様々な改良が加えられて大規模データを扱え、機能も大きく拡張させている。
HyenaDNAは、HyenaというTransformer並みの性能は保ちながら既存のLLMが抱えていた入力長の二乗で計算コストがふえるという問題を解決する深層学習モデル(大域的な畳み込みフィルターとデータ制御ゲート機構を用いる)を遺伝子解析に適用するモデルである。これで、ヌクレオチド単位での解像度で100万のシーケンスの処理が可能となった。
2024年11月に発表された最初のEvoモデルは、DNA、RNAとタンパク質という広い対象に対してつながっている配列を明らかにするという目標をかかげて登場した。270万件の原核生物とバクテリオファージのゲノム、3000億のヌクレオチドのトークンで学習させた単一ヌクレオチドの解像度を持つ。
コアのアーキテクチャーはStripedHyenaで、70億パラメータでHyenaの改良版で長い配列を扱える新しいタイプの深層学習を使っており、トランスフォーマーで使われるような伝統的なアテンションメカニズムとは異なる。131,072トークンもの長いコンテキストを処理できる。
配列の解明に加え、ゲノム規模の配列を新たに作ったり、ゲノム全体にわたる遺伝子の性質を予測(たとえば突然変異がタンパク質やRNA、DNAにどのような影響を与えるか)をゼロショットでできる。
これが、2025年2月に発表されたEvo2では、更にバイオインフォマティックスを幅広くカバーする大規模言語モデルとして発表された。DNA、RNA、タンパク質の解明を、真核生物、原核生物、古細胞にわたって行える。アーキテクチャーは更にStripedHyena 2に進化し、400億個のパラメータ、100万トークンのコンテキストで処理できる。
8.85兆のヌクレオチド、15,032の真核生物のゲノム、113,379の原核生物のゲノムで学習しており、異種生物間での一般化や対象生物も広がっている。
マルチオミクスは成果が出始めるのはまだ先だと思われるが、様々な試みがはじまった。
(参考情報)
1.AI is helping the Healthcare Industry Write the Next Chapter in Medicine
GTC2025(2025/3フィジカル・オンライン同時開催)でのNvidiaのヘルスケア領域の責任者のVP、Kimberly Powell氏の講演。Nvidiaのヘルスケア対応の紹介をしている。非常によくまとまった内容。Evo2もごく簡単に紹介。
https://www.nvidia.com/en-us/on-demand/session/gtc25-s71353/
2.Understanding the Language of Life’s Biomolecules Across Evolution
at a New Scale with Evo 2 Feb 19, 2025 By Kyle Tretina
NvidiaのEvo2に関する発表論文。
https://developer.nvidia.com/blog/understanding-the-language-of-lifes-biomolecules-across-evolution-at-a-new-scale-with-evo-2/
3.Genome modeling and design across all domains of life with Evo 2
Evo2を発表した論文で2025/2/21にポスティングされているがレビューは終わっていないと注意書きがある。
https://www.biorxiv.org/content/10.1101/2025.02.18.638918v1 からダウンロード可能
4.DNA language model: Evo2の基礎と応用
AIのエンジニアで仕事でもライフサイセンス領域を専門としているkamata氏によるEvo2の解説記事で2025/6/16にnoteに掲載された。技術的にも詳しく非常に内容が濃い。
https://note.com/olachin/n/n08e15b12afd2
5.Large Language Models in Bioinformatics: A Survey
バイオインフォマティックス分野でのLLMを整理した論文の中で新しいもの。
(2025/5にポスティングされ2025/7のACL 2025で発表されたもの)
https://aclanthology.org/2025.findings-acl.184/
- PROFILE
-

柴柳 健一
大手ITベンダーでの海外ビジネス、アライアンス事業の経験を活かし米国最先端ICT技術の動向調査、コンサルを行っている。
- CATEGORY
